记录一下最近了解的聚类算法:DBSCAN
DBSCAN is a density-based clustering algorithm that groups together data points that are closely packed, while marking points that lie alone in low-density regions as outliers.
Unlike K-means, DBSCAN doesn't require specifying the number of clusters beforehand and is robust to noise and outliers. Along with its hierarchical extensions HDBSCAN, it is applied mostly in Automotive Radar Data.
最近对有关雷达数据的聚类方法做了些调研,主要看的一些评估指标和有关DBSCAN的方法,于此重新复习一下DBSCAN。文件附上: pdf文件
和K-Means,BIRCH这些一般只适用于凸样本集的聚类相比,DBSCAN既可以适用于凸样本集,也可以适用于非凸样本集。下面我们就对DBSCAN算法的原理做一个总结。
凸集 (Convex Set):对于该集合中的任意两点,连接它们的线段��上的所有点也都包含在该集合中。
凹集 (Concave Set):对于该集合中的任意两点,连接它们的线段上的所有点也都不在该集合中。
Clustering
聚类算法主要有 4 大类:
- Centroid-based Clustering
基于质心的聚类从许多随机定位的质心(中心点)开始,这些质心充当所找到簇的中心。这些质心被一步步细化,直到它们收敛到簇的真正中心。 - Density-based Clustering
基于密度的聚类组按其附近其他点的密度进行分组。真正靠近的点将形成一个簇,而孤立的点将被标记为异常值 - Distribution-based Clustering
基于分布的聚类首先假设每个聚类都由从已知分布(通常是正态分布)中采样的点组成。该算法的目标是找到每个聚类的分布参数 - Hierarchical Clustering
很多情况下的簇并不像我们看到的示例那样清晰地分开。它们通常由较小的组组成,这些组可以组合在一起形成一个更大的簇,最终得到一个包罗万象的簇。
DBSCAN基本概念
DBSCAN的基本概念可以用1,2,3,4来总结。
-
1个核心思想:基于密度。直观效果上看,DBSCAN算法可以找到样本点的全部密集区域,并把这些密集区域当做一个一个的聚类簇。
-
2个算法参数:邻域半径R和最少点数目MinPoints。这两个算法参数实际可以刻画什么叫密集:当邻域半径R内的点的个数大于最少点数目MinPoints时,就是密集
-
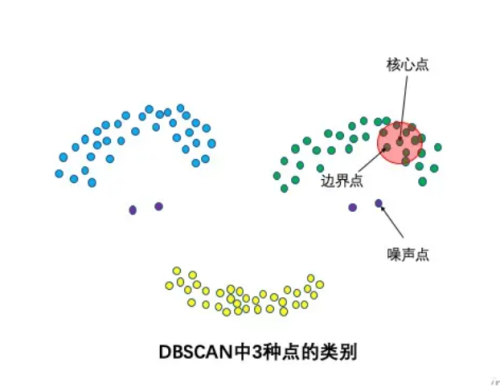
3种点的类别:核心点,边界点和噪声点。邻域半径R内样本点的数量大于等于minpoints的点叫做核心点。
不属于核心点但在某个核心点的邻域内的点叫做边界点。既不是核心点也不是边界点的是噪声点
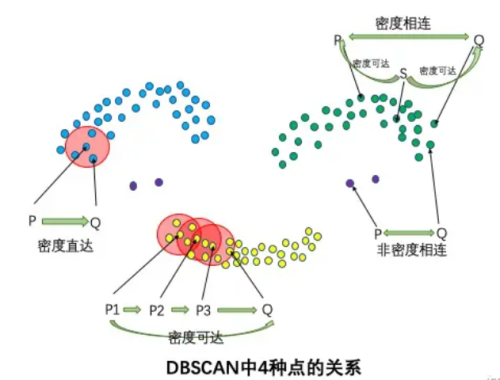
- 4种点的关系:密度直达,密度可达,密度相连,非密度相连。
如果P为核心点,Q在P的R邻域内,那么称P到Q密度直达。任何核心点到其自身密度直达,密度直达不具有对称性,如果P到Q密度直达,那么Q到P不一定密度直达。
如果存在核心点P2,P3,……,Pn,且P1到P2密度直达,P2到P3密度直达,……,P(n-1)到Pn密度直达,Pn到Q密度直达,则P1到Pn(n=1,2...),Q密度可达。密度可达也不具有对称性。
如果存在核心点S,使得S到P和Q都密度可达,则P和Pn(n=1,2...),Q密度相连。密度相连具有对称性,如果P和Q密度相连,那么Q和P也一定密度相连。密度相连的两个点属于同一个聚类簇。
如果两个点不属于密度相连关系,则两个点非密度相连。非密度相连的两个点属于不同的聚类簇,或者其中存在噪声点
DBSCAN聚类思想
DBSCAN的聚类定义很简单:由密度可达关系导出的最大密度相连的样本集合,即为我们最终聚类的一个类别,或者说一个簇。
这个DBSCAN的簇里面可以有一个或者多个核心对象。如果只有一个核心对象,则簇里其他的非核心对象样本都在这个核心对象的ϵϵ-邻域里;如果有多个核心对象,则簇里的任意一个核心对象的ϵϵ-邻域中一定有一个其他的核心对象,否则这两个核心对象无法密度可达。这些核心对象的ϵϵ-邻域里所有的样本的集合组成的一个DBSCAN聚类簇。
那么怎么才能找到这样的簇样本集合呢?DBSCAN使用的方法很简单,它任意选择一个没有类别的核心对象作为种子,然后找到所有这个核心对象能够密度可达的样本集合,即为一个聚类簇。接着继续选择另一个没有类别的核心对象去寻找密度可达的样本集合,这样就得到另一个聚类簇。一直运行到所有核心对象都有类别为止。
但是我们还是有三个问题没有考虑。
第一个是一些异常样本点或者说少量游离于簇外的样本点,这些点不在任何一个核心对象在周围,在DBSCAN中,我们一般将这些样本点标记为噪音点。
第二个是距离的度量问题,即如何计算某样本和核心对象样本的距离。在DBSCAN中,一般采用最近邻思想,采用某一种距离度量来衡量样本距离,比如欧式距离。这和KNN分类算法的最近邻思想完全相同。对应少量的样本,寻找最近邻可以直接去计算所有样本的距离,如果样本量较大,则一般采用KD树或者球树来快速的搜索最近邻。
第三种问题比较特殊,某些样本可能到两个核心对象的距离都小于ϵϵ,但是这两个核心对象由于不是密度直达,又不属于同一个聚类簇,那么如果界定这个样本的类别呢? 一般来说,此时DBSCAN采用先来后到,先进行聚类的类别簇会标记这个样本为它的类别。也就是说BDSCAN的算法不是完全稳定的算法。
DBSCAN算法设计
DBSCAN(DB, distFunc, eps, minPts) {
C := 0 /* Cluster counter */
for each point P in database DB { /*对数据集中的每个点处理*/
if label(P) ≠ undefined then continue /* Previously processed in inner loop */
Neighbors N := RangeQuery(DB, distFunc, P, eps) /* Find neighbors */
if |N| < minPts then { /* Density check */
label(P) := Noise /* Label as Noise */
continue
}
C := C + 1 /* 若满足簇数+1 */
label(P) := C /* 用集群标签`C`标记当前点`P`。 */
SeedSet S := N \ {P} /* 我们初始化一个种子集 S ,其中包含 P 除其自身之外 P 的所有邻居。 */
for each point Q in S { /* Process every seed point Q */
if label(Q) = Noise then label(Q) := C /* Change Noise to border point */
if label(Q) ≠ undefined then continue /* Previously processed (e.g., 如果是边界点) */
label(Q) := C /* 用集群标签C标记当前邻居点 */
Neighbors N := RangeQuery(DB, distFunc, Q, eps) /* Find point Q's neighbors */
if |N| ≥ minPts then { /* 如果点Q的邻居数|N|大于或等于minPts,则表示它是核心点 minPts*/
S := S ∪ N /* Add new neighbors to seed set */
}
}
}
}
DBSCAN小结
优点
- 不需要先验地指定数据中的簇数。
- DBSCAN可以找到任意形状的簇。
- DBSCAN 具有噪声的概念,并且对异常值具有鲁棒性。
- DBSCAN设计用于可以加速区域查询的数据库,例如使用R*树。
缺点
- DBSCAN 的质量取决于函数
regionQuery(P,ε)中使用的距离测量值。最常用的距离度量是欧几里得距离。特别是对于高维数据,由于所谓的“维诅咒”,这个指标几乎毫无用处,很难找到合适的ε值。然而,这种效应也存在于任何其他基于欧几里得距离的算法中。 - DBSCAN 在处理包含不同密度聚类的数据集时会遇到困难。它假设团簇具有均匀的密度,并且可能无法识别具有显着不同局部密度的团簇。
- 在最坏的情况下,DBSCAN的时间复杂度O(n^2),即数据点的数量。对于大型数据集,可能很费时间,难以保证实时性。
- DBSCAN 无法很好地聚类密度差异较大的数据集,因为无法为所有聚类适当选择
minPts-ε组合
DBSCAN扩展
Generalized DBSCAN (GDBSCAN) is a generalization by the same authors to arbitrary "neighborhood" and "dense" predicates.
The ε and minPts parameters are removed from the original algorithm and moved to the predicates. For example, on polygon data, the "neighborhood" could be any intersecting polygon, whereas the density predicate uses the polygon areas instead of just the object count.
Various extensions to the DBSCAN algorithm have been proposed, including methods for parallelization, parameter estimation, and support for uncertain data. The basic idea has been extended to hierarchical clustering by the OPTICS algorithm. DBSCAN is also used as part of subspace clustering algorithms like PreDeCon and SUBCLU.
HDBSCAN* is a hierarchical version of DBSCAN which is also faster than OPTICS, from which a flat partition consisting of the most prominent clusters can be extracted from the hierarchy. Reference